2022.10.21改訂(2020.4.9, 2012.4.22)
解析例トップ
医薬品・化粧品トップページ
化粧品設計システム、FFEのユーザーが面白い論文を送ってきてくれた。
International Journal of Engineering Science and Technology Vol. 2(6), 2010, 1526-1531
Prediction of Skin Penetration using Artificial Neural Network
化合物の皮膚への浸透をニューラルネットワークを用いて検討したものだ。
論文中では以下の化合物の透過係数、Kp(cm/h)を用いていた。
データをコピペして自分でもやってみよう。
透過係数と構造
| HCode | CAS | Name | SMILES | logKp |
|---|---|---|---|---|
| 46 | 62-53-3 | aniline | NC1=CC=CC=C1 | -2.65 |
| 48 | 100-66-3 | anisole | COC1=CC=CC=C1 | -1.6 |
| 52 | 71-43-2 | benzene | C1=CC=CC=C1 | -0.9547 |
| 58 | 100-51-6 | benzyl alcohol | OCC1=CC=CC=C1 | -2.22 |
| 325 | 64-17-5 | ethyl alcohol Ethanol | CCO | -3 |
| 490 | 108-11-2 | 4-methyl-2-pentanol | CC(C)CC(C)O | -2.33 |
| 904 | 120-80-9 | pyrocatechol | C1=CC=C(C(=C1)O)O | -2.77 |
| 1071 | 123-07-9 | p-ethylphenol | CCC1=CC=C(O)C=C1 | -1.46 |
| 1200 | _58-08-2 | Caffeine | O=C(N(C)C2=C1N(C)C=N2)N(C)C1=O | -4 |
| 1204 | 69-72-7 | salicylic acid | OC1=CC=CC=C1C(O)=O | -2.2 |
| 5291 | 106-44-5 | p-cresol | OC1=CC=C(C)C=C1 | -1.76 |
| 9291 | 767-00-0 | Paraoxybenzonitrile | N#Cc1ccc(O)cc1 | -1.983 |
| 20324 | 15307-86-5 | Diclofenac | C1=CC=C(C(=C1)CC(=O)O)NC2=C(C=CC=C2Cl)Cl | -1.7399 |
| 20333 | 132-22-9 | Chlorpheniramine | CN(C)CCC(C1=CC=C(C=C1)Cl)C2=CC=CC=N2 | -2.66 |
| 20448 | 50-22-6 | Corticosterone | C[C@]12CCC(=O)C=C1CC[C@@H]3[C@@H]2C@HO | -4.22 |
| 21376 | 76-57-3 | Codeine | O[C@H]2C=C/[C@H]5[C@@H]4N(CC[C@@]51c3c(O[C@H]12)c(OC)ccc3C4)C | -4.3098 |
| 22213 | 57-43-2 | 5-Ethyl-5-(3-methylbutyl)-barbituric acid {Amobarbital} | O=C1NC(=O)NC(=O)C1(CCC(C)C)CC | -2.644 |
| 22239 | 77-28-1 | 5-Butyl-5-ethylbarbituric acid {Butobarbital} {Butetal} | O=C1NC(=O)NC(=O)C1(CCCC)CC | -3.7144 |
| 977 | 615-74-7 | 2-Chloro-5-Methyl Phenol | OC1=C(Cl)C=CC(C)=C1 | -1.26 |
| 22346 | 88-04-0 | 4-Chloro-3,5-xylenol | Oc1cc(C)c(Cl)c(C)c1 | -1.2774 |
幾つかの化合物については馴染みの無い構造だろう。それらの構造は次のようになる。
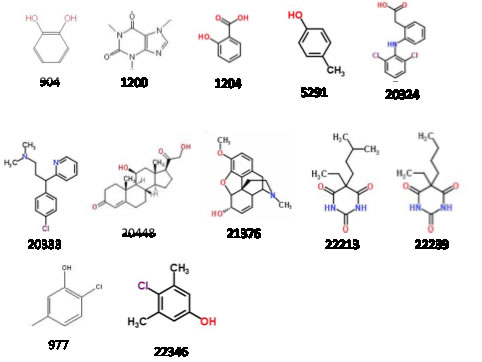
持っている官能基も分子のサイズもとても異なっているのが分かるだろう。
論文ではAbraham の識別子、R2 (excess molar refraction), π2 H (dipolarity/polarizability), Σα2 H, Σβ2 H (the overall or effective hydrogen-bond acidity and basicity), と Vx (the McGowan characteristic volume)を使ってニューラルネットワークを使って学習させて、学習に使わなかった化合物について予測を行いモデルの検証を行っている。
これをHansenの溶解度パラメータ(HSP)を使ってやってみよう。
HSPiP のQSARツール
HSPiPのver.5からQSARツールが付属している。
先ほどのテーブルを表計算ソフトにペーストしておく。
表計算ソフトから、化合物の名称、Smilesの構造式、logKpをクリップボードにコピーする。(Ctrl-C, Command-C)
そして、メインプログラムからQSARボタンをクリックする。そしてクリップボードからペーストボタンをクリックすると先ほど選択したテーブルがQSARパネルにペーストされる。
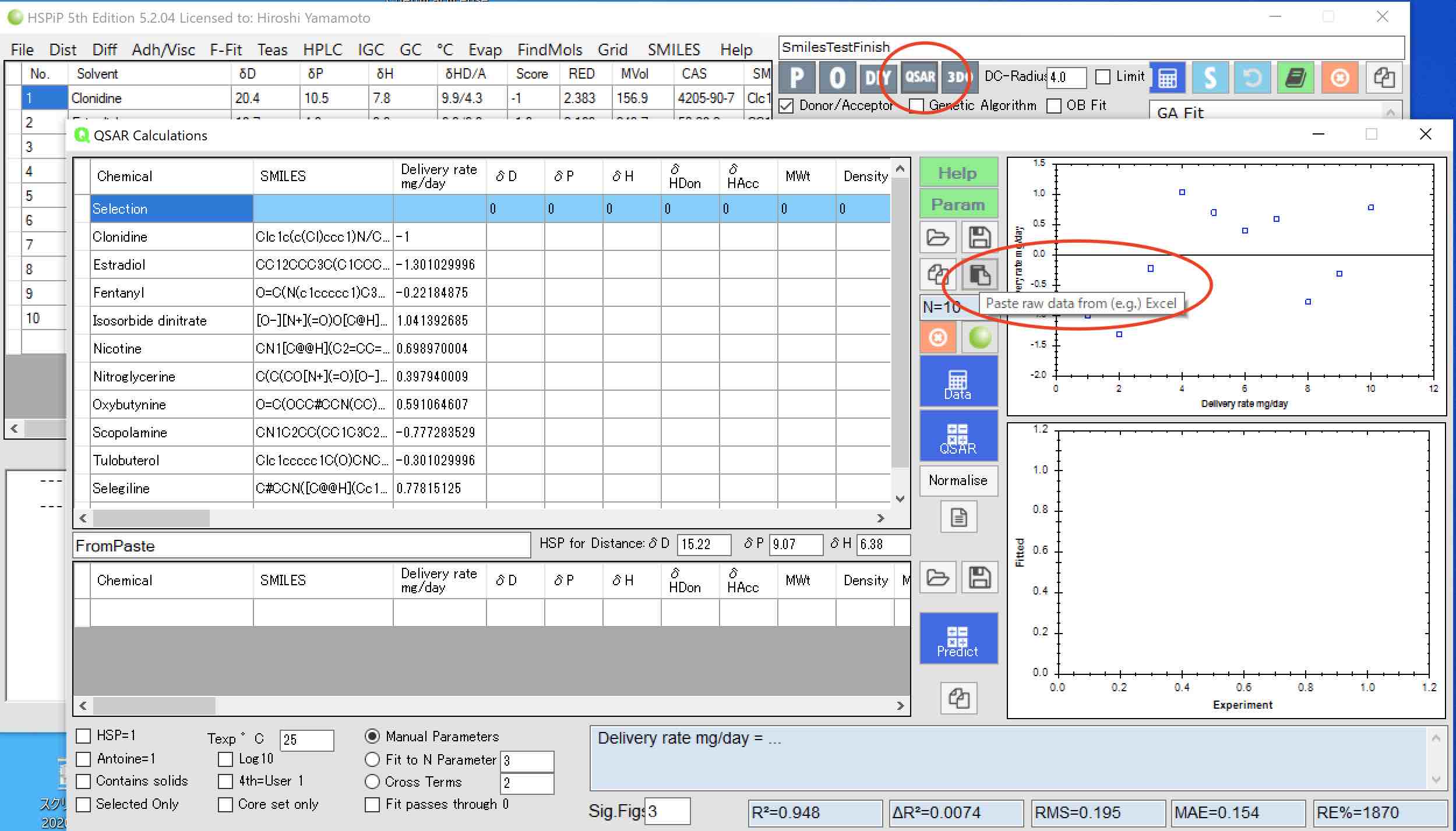
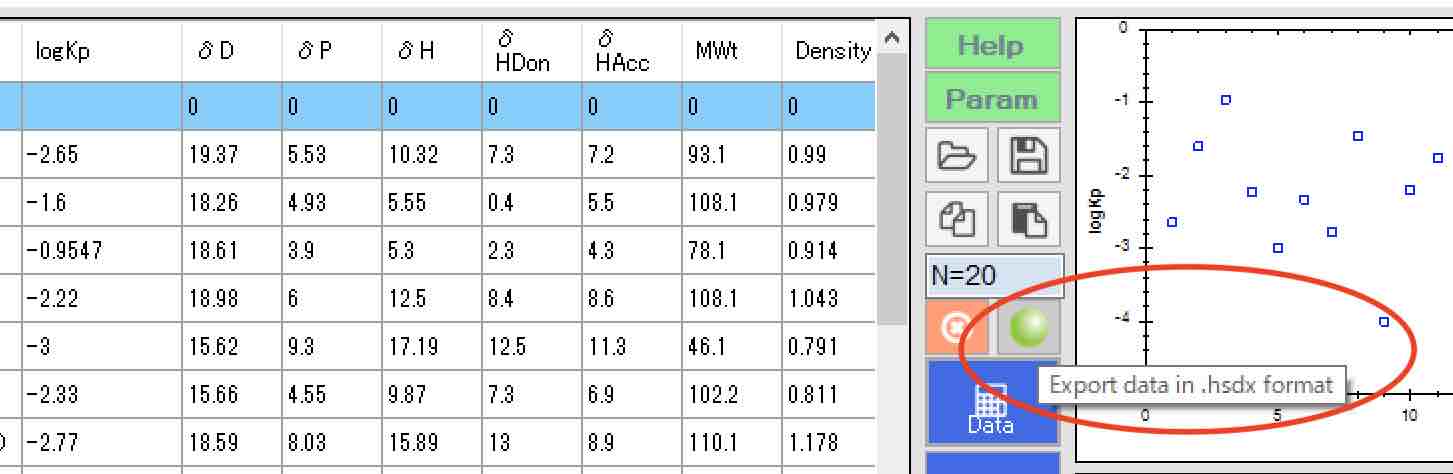
Dataボタンをクリックすると、Smilesの構造式から様々な物性値を推算し,テーブルが埋まる。そこまでできたら、データをhsdxフォーマットにエクスポートする。
メインプログラムから、セーブしたhsdxファイルを読み込む。
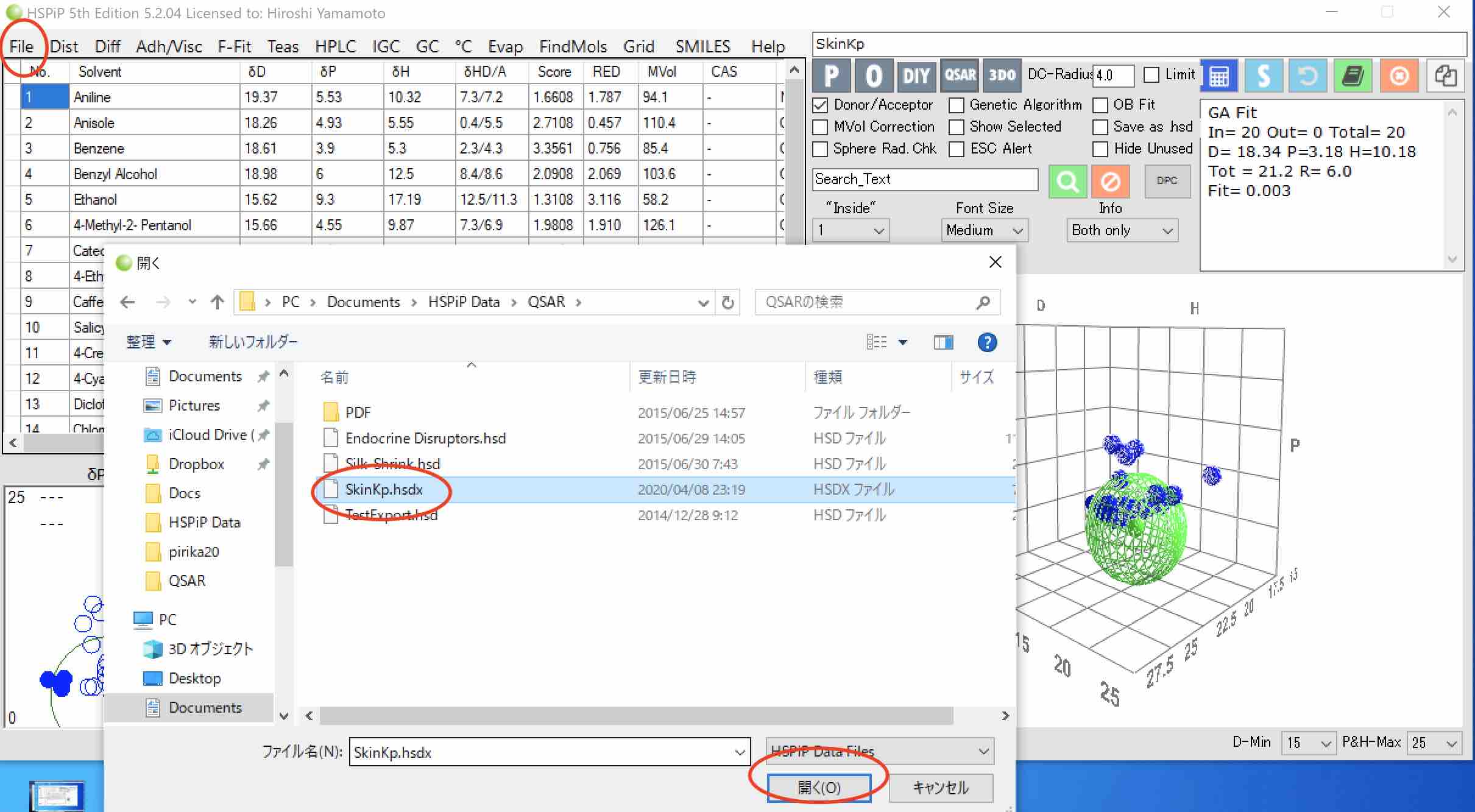
これでHSPiPでSphereを計算する準備が整う。
Scoreには自動的にlogKpの値が入力されている。
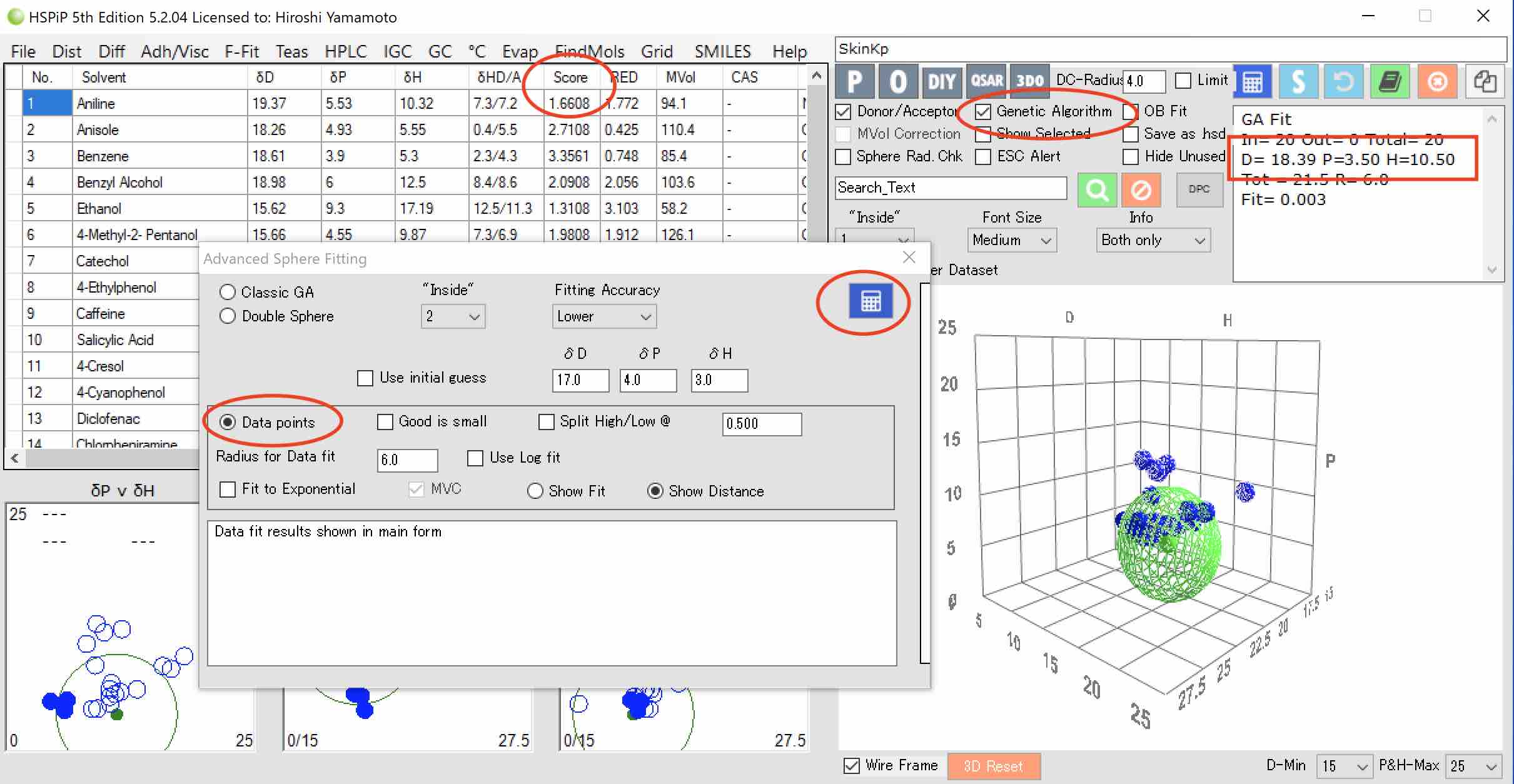
Scoreが実数の場合には、Genetic Algorithm(GA)オプションをonにして、Data Point(定量的なSphere)を選択しSphereを計算する。
GUIは頻繁に更新されていて、ver. 5.4ではSphere計算関係のオプションは左下にまとめられている。
(簡単に言えば、Kpの値が大きいものは皮膚への溶解度が高いということだ。皮膚のHSPをある値に仮定し、皮膚と化合物のHSP距離が短いものはKpが大きく、距離が長いものほどKpが小さくなるように皮膚のHSPを決定する。)
皮膚のHSPは [18.39, 3.5, 10.50]となった。
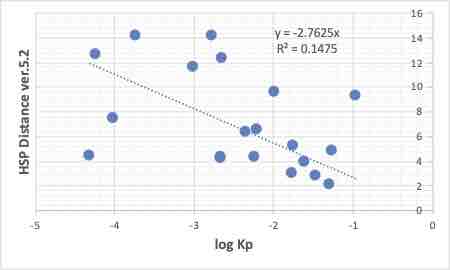
化合物と皮膚のHSP距離は、上図に示すように、非常に低い相関になる。
HSP距離=(4*(dD-18.39)^2 + (dP-3.5)^2 + (dH-10.5)^2 )^0.5
Drag=回転, Drag+Shift キー=拡大、縮小, Drag+コマンドキーかAltキー=移動。
溶媒をクリックすれば溶媒の名前が現れる。
緑色の大きな球が皮膚を表し、赤と青の球が今回の試験体になる。
赤い球で示したものが、Kpが大きなもの、青い球がKpが小さなものを示している。
皮膚のHSPと赤い球の距離が短くないので、これはおかしいと判るだろう。
実に、この計算ではHSPは水素結合項をdHacid, dHbaseと分割して考慮しないと正しい距離を示さない。
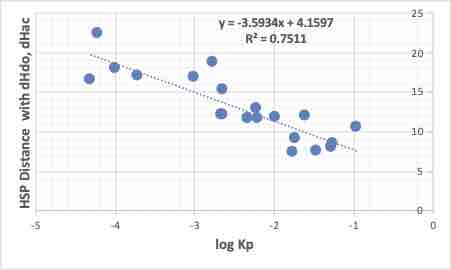
HSP距離=(4*(dD-17.06)^2 + (dP-3.03)^2 + (dHAcid-9.4)^2 +(dHbase-0)^2 )^0.5
皮膚のHSP=[dD, dP, dHacid, dHbase]=17.06, 3.03, 9.4, 0]になる。
しかし、ドナー/アクセプターに分割すると4次元ベクトルになり、それを3次元にプロットすると、このようにおかしな表示になってしまう。
非常に悩ましく開発者も困っている宿題だ。
dHacid,dHbaseを含むHSP距離
2020.4.9
HSPiPではdHdo,dHacと表記しているが、これは本当はdHacid, dHbaseと表記すべきものだ。
また、dHをdHacid,dHbaseに分割した場合の、HSP距離については未だに確定はしていない。
思考実験として、ある化合物が、例えば、酢酸によく溶解したとする。
それは、ターゲット化合物中にカルボキシル基があって、(dHacid1 – dHacid2)^2 が0に近く、HSP距離が短くなって溶解すると考えるか?
もしくは、ターゲット化合物中にアミンを持ち、(dHacid1 – dHacid2)*(dHbase1 – dHbase2)で酸-塩基相互作用で溶解が促進されるか?
どちらであるかはわからない。
本来のドナー/アクセプター相互作用では、(dHacid1 – dHacid2)*(dHbase1 – dHbase2)を使うべきだが、これは場合によるとHSP距離のルートの中がマイナスになるという不都合が生じる事がある。
そこで、山本の個人的見解だが、
HSP距離=(4*(dD-17.06)^2 + (dP-3.03)^2 + (dHacid-9.4)^2 +(dHbase-0)^2 )^0.5
タイプのHSP距離(>0)を採用する。
この距離を求める機能はHSPiPには搭載されていない。
続き
文献を送ってくれた方が面白いアイデアを送ってくれた。(2012.4.24)
4次元を3次元に落とす際に、dDとdPをまとめてしまってdDPとdHacid, dHbaseの3次元で表示すればいいというものだ。
もともと、今の表示でも、dH=sqrt(dHacid^2 + dHbase^2 )として4次元を3次元に落としている。「HSP距離を見たいのであれば、dDP= sqrt(4*dD^2 + dP^2 )としてしまえば正しい距離を表示するはずだ」というものだ。
(ついでに試験体の分子体積を溶媒球のサイズに指定することもできます。距離が小さいのにKpが小さいものは分子が大きいことがわかる。)
このdHacid, dHbaseを含む4D HSPの取り扱いにはHSPiPだけでは今のところ不十分だ。
ここで注意して欲しい点は、これは皮膚のHSPベクトルと試験体のHSPベクトルだけでここまで説明できてしまう事だ。
分子のサイズは入っていない。
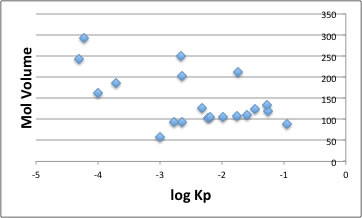
通常こうした検討は、オクタノール/水分配比率 logKowで評価する事も多いだろう。
しかし、logKpとlogKowをプロットすると下の図のように余り相関があるとは言え無い。
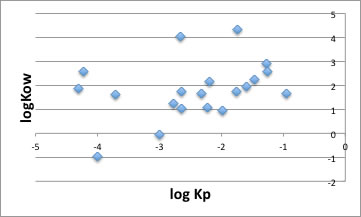
分子体積の効果も、分子が大きいとlog Kp も小さくなる傾向があるが、分子が大きくてもlog Kpが大きいものもあり、分子体積が同じ100程度でもKpの値は100倍異なるものがある事も分かる。
これらの指標に対してハンセンの溶解度パラメータを用いた方法は非常に明確に現象を説明している事がわかる。
つまり、”似たものは似たものを溶かす”だ。
私達は、ここで言う似たものとは、HSP[dD, dP, dH]のベクトルが似たものは、ベクトルが似たものを溶かすと表現する。
それでは透過係数に影響を与える効果としてはHSP以外どんなものがあるだろうか?
Y-MBの吐き出す推算結果を利用して、QSAR式の構築を試みてみよう。
このQSARというのは、定量的構造活性相関の事で、物理化学的な意味合いはほとんどない(単なるフィティングである)が、どのような識別子が相関を上げるのかを検討するには有効だ」。
識別子の組み合わせを相関係数が高くなるように自動的に探索するプログラムを利用して変数選択を行うと、一番重要な変数は当然HSP距離だが2番目に重要な変数は水への溶解度の対数をとった、logSだった。
2つを組み合わせて作ったモデル式は下に示すように、決定係数R^2 =0.83476となる。
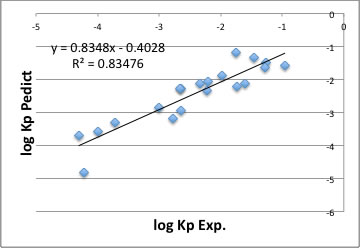
log KP = A*HSP距離 + B*logS + C
どちらのパラメータがどのぐらい効いているかを表すA, B, Cは自分で計算してみよう。
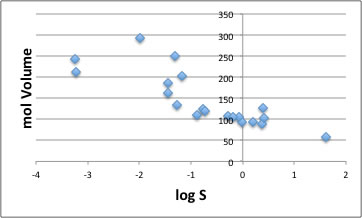
何故QSARを使っても分子体積の項目は選ばれなかったのだろうか?
それはlogSと分子体積は下の図のようにかなり高い相関があるからだろう。
つまり、log Sは、分子体積の効果も取り込み、水への溶解度の効果も含んだパラメータであるので、それとHSP距離を組み合わせたQSAR式が最も効果的だとプログラムが判断した事になる。
それでは、logSは皮膚透過においてどのような意味合いを持つのだろうか?
Yakugaku Zasshi 13283) 319-324 (2012) ,木村、藤堂、杉林らの論文、”ナノ粒子の皮膚浸透性と安全性”に示唆に富んだ説明があった。
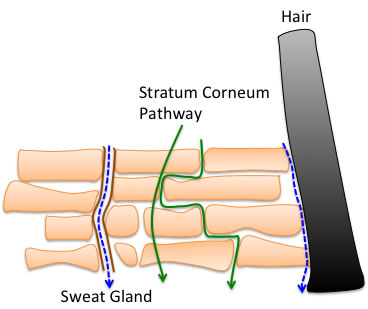
化学物質が経皮吸収されるには、化学物質が溶媒(媒質)に溶解し、そこから、角層(主に角質細胞内脂質)へ分配し、表皮を拡散していく。
おそらく、その部分はHSPの溶解度パラメータが支配的なのだろう。
しかし、それ以外にも汗腺などの付属器官ルートがあり、それがlogSに相当する部分、透過を助けているのではないかと考えられているようだ。
さらに多くの皮膚透過のデータを集めていただいたのでさらに詳しい解析を行おうと思う。
HSPiPのQSAR機能
HSPiPにもQSAR機能が搭載されているし、同じ例題がサンプルとして付属している。しかし、HSP距離が[dD,dP,dH]でしか取れないため、上記のような結果にはならない。試しにどのようなものが選ばれるかやってみよう。
QSAR画面から、ファイルオープンをクリックして、SkinPenetration3.hsqを選ぶ。
Fit to N Parameters 2としてQSAR式を構築する。
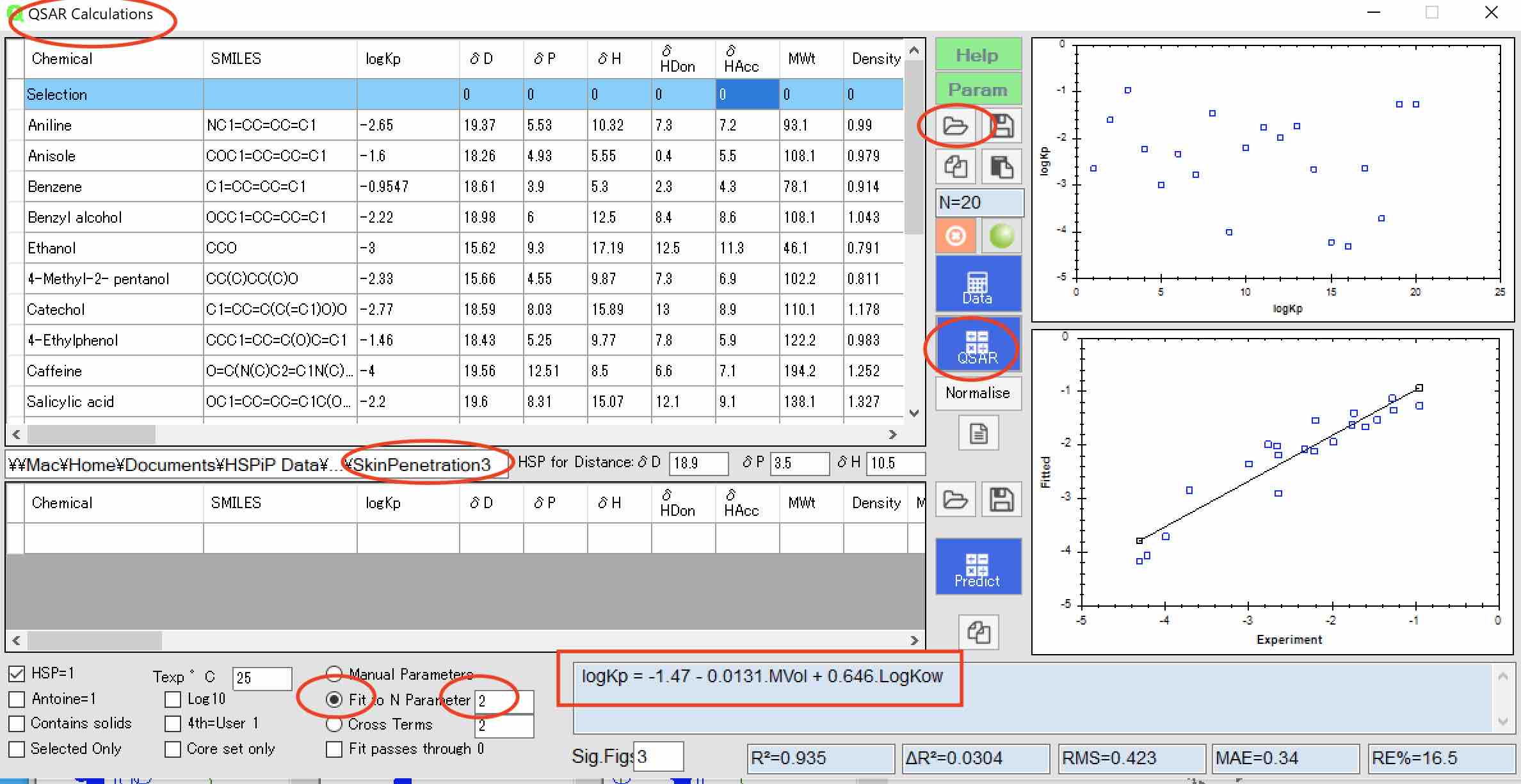
logKp = -1.47 – 0.0131.MVol + 0.646.LogKow
決定係数R^2 =0.935という良好なモデルを作成する事ができた。
こうした結果を見ると、世間一般で、logKowの重要性が強調される理由がわかると思う。
logKowが分配係数で、100/100でも0.01/0.01でもlogKowは同じになってしまう事から、このパラメータを嫌う研究者も多くいる。
私の立場は、系統の異なる様々なQSAR式を作成して、多面的に検討しましょうという事だ。
MO計算結果、RDKitなどのトポロジカルなインデックス使えるものは何でも使って複数の式を構築していき、総合判断するのが大事だろう。
主成分分析(PCA)の利用
2022.10.21
昔は、多次元ベクトルを次元縮退させるのにSOM(Self Organization Map)を使うことが多かった。
最近は主成分分析(PCA)を行うことが多い。
[dD, dP, dHacid, dHbase]の4次元をPCAを使い3次元に縮退した。
解析例トップ
医薬品・化粧品トップページ
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください
