2021.1.8
その他の化学トップページ >
JSMEで分子を描いて3次元の分子構造を取り出そう
JSME-RDKit (2020.12.17)を別タブで開く
JSMEで分子を描いてGet 3D-Structureボタンをクリックすると、JSMEからSmilesの構造式が作成されます。そして、RDKitを用いて、Smilesの構造式から3次元の分子が組み立てられ、テキストエリアに分子の3次元構造が出力されます。
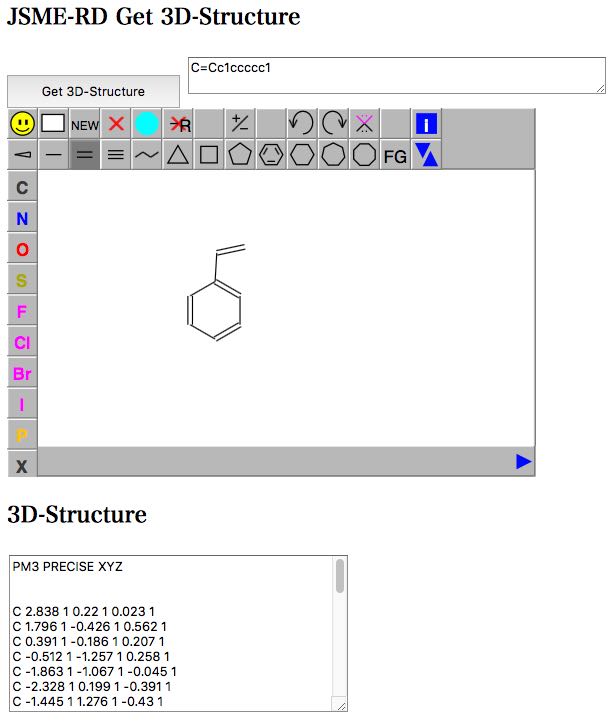
この分子構造のフォーマットは、MOPAC用の入力ファイルになっています。
ファイルの後ろに、Bond Info.(結合情報)が出力されています。
MOPACの計算自体には必要無いので消去してください。
ここで作られた、構造を実際に表示させて見ると次のようになります。
2重結合と、ベンゼン環が同じ平面に無いことがわかります。
RDKitがSmiles構造式から3次元構造を作成する際に、分子力学(Molecular Mechanics)計算を行い、構造を最適化させます。その際の共役の取り方の問題で、自動的に構造生成させる以上、どうしようも無いことです。
エチレンが2重結合の真ん中でねじれたような構造は、取り出せません。
そのような時には、Y-Molで修正を行います。
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください。
