2021.1.29
その他の化学トップページ >
Smiles構造式から3次元の分子構造を取り出そう
RDKit Smiles to 3D-Structure (2021.1.29)を別タブで開く
PubChemなどで分子を検索すると直ぐにSmilesの構造式が得られます。そして、RDKitを用いて、3次元の分子が組み立てられ、テキストエリアに分子の3次元構造が出力されます。
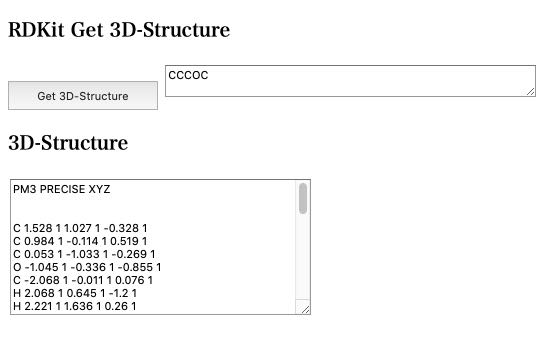
この分子構造のフォーマットは、MOPAC用の入力ファイルになっています。
ファイルの後ろに、Bond Info.(結合情報)が出力されています。
MOPACの計算自体には必要無いので消去してください。
Smilesの構造式から、2次元の分子(SVG, png)を見ながら、3次元構造を入手したのでしたら次のリンクへ進んでください
RDKit Smiles to 2D & 3D-Structure (2021.1.29)を別タブで開く
例えば、RemdesivirのSmilesを入れると、次のように2次元の分子を表示した上で、3次元の構造も取り出すことができます。Pngに変換ボタンを押すと、Png画像が表示されるので、右クリックで画像を取り出す事ができる。
Nc1ncnn2c1ccc2C3(C#N)C(O)C(O)C(O3)COP(=O)(Oc4ccccc4)NC(C)C(=O)OCC(CC)CC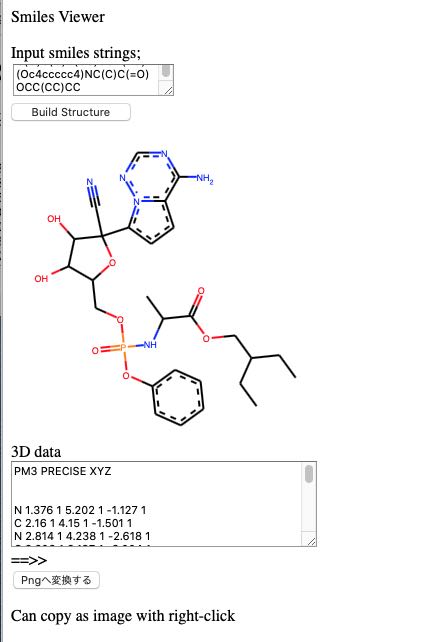
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください。
