2021.9.20
情報化学+教育 > MAGICIAN 養成講座 > マイクロ波と3つのMI > 有機物の識別子の作成法
MAGICIAN(MAterials Genome/Informatics and Chemo-Informatics Associate Networks)
MAGICIANとは、材料ゲノム(Materials Genome)、材料情報学(Materials Informatics)、情報化学(Chemo-Informatics)を結びつけて(Associate)ネットワーク(Networks)を構築していかれる人財です。
その1、データ収集のデータは表計算ソフトにペーストしてありますか?
ここでは、有機物のデータに識別子を加えて行きます。
データソース
次のWebアプリを使って識別子を作成します。
- YMB(熱力学的物性値推算ソフトウエアー)。
- RDKit(トポロジカル・インデックス)
- CNDO/2 分子軌道計算
YMBによる識別子の作成に関しては、元は、pirikaの物性推算のページにあります。ここで取り扱っている物性値に関してGUI(グラフィカル・ユーザー・インターフェイス)を付け加えたものがYMBになります。
第1世代のYMBに関しては、こちらを参照してください。
第2世代のYMBに関しては、一時期、HSPiPに搭載された、Y-Predictが原型になります。
大学で授業に用いているYNU-YMBは、ボタンなどの形状が少し異なりますが、基本は第2世代になります。(YNU-YMBは一般公開はしていません。学生と一部の人柱だけが使えます。)
RDKit自体はオープン・ソースのライブラリーです。使い方はネットの上に溢れていますので、それらを参照してください。
どんなトポロジカルな情報を吐き出すのかは、マニュアルに記載されています。
RDKitはPythonから扱うのが普通で、それが原因で利用を躊躇うことも多いかもしれません。Pirikaではブラウザーから計算できるようにしてあります。つまりSmilesの構造式が準備されているなら、後はブラウザーだけでトポロジカル・インデックスを計算する事ができます。
CNDO/2の分子軌道計算に関しては、おおもとは、pirikaのこちらのページを参照してください。
YMB-識別子の作成
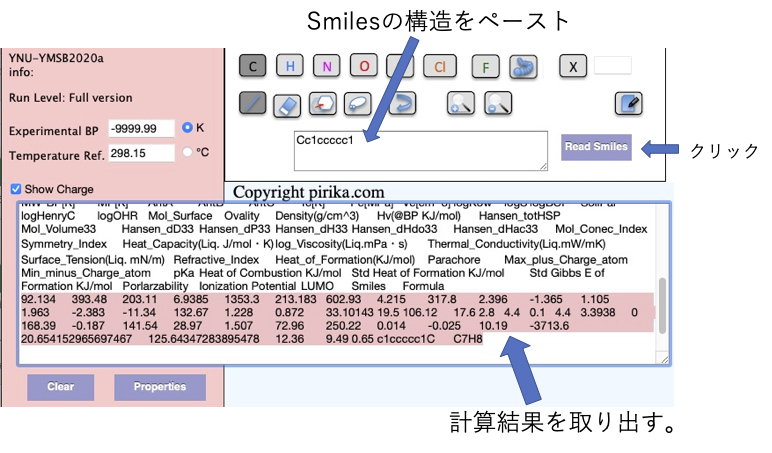
作成方法はとても簡単で、Smilesの構造式をテキストエリアに貼り付け、Read Smilesボタンをクリックするだけです。
計算結果は、最初の計算の時はタイトルも含めてコピペします。
Smilesのリストを一括で計算するProバージョンもありますが、学生はSmilesどころかきちんと分子構造を描くところからやってください。どんな官能基がどんな物性を持つのか体感し、化学の常識を身につけることも大事です。
簡単な分子の描き方は[こちらを参照]()してください。
YMBは一般公開してないので、結果だけをまとめておくと次のようになります。
YMBはSmilesの構造式から次の物性を予測します。
MW 分子量
BP[K] 沸点
MP[K] 融点
AntA AntB AntC アントワン定数
Tc[K] Pc[MPa] Vc[cm^3] 臨界定数
logKow オクタノール/水分配比率
logS [g/100g水] 水への溶解度
logBCF 生物濃縮係数
SoilPar 土への分配係数
logHenryC ヘンリー定数
logOHR OHラジカルとの反応速度
Mol Surface (Å^2 ) Mol Volume (Å^3 ) 分子表面積、体積
Ovality 球形度
Density(g/cm^3) 密度
Hv(@BP KJ/mol) 沸点における蒸発潜熱
Hansen totHSP ハンセン溶解度パラメータのトータル
Hansen dD Hansen dP Hansen dH HSP,分散項、分極項、水素結合項
Hansen dHAcid Hansen dHbase 水素結合項Acid, Base
Mol Conec Index 分子結合インデックス
Symmetry Index 対称性インデックス
Heat Capacity(Liq. J/mol・K) 気体の熱容量
log Viscosity(Liq.mPa・s) 液体の粘度
Thermal Conductivity(Liq.mW/mK) 熱伝導度
Surface Tension(Liq. mN/m) 表面張力
Refractive Index 屈折率
Heat of Formation(KJ/mol) 生成熱
Parachore パラコール
Max plus Charge_atom 最大+電荷
Min minus Charge atom 最小-電荷
pKa pKa
Heat of Combustion KJ/mol 燃焼熱
Std Heat of Formation KJ/mol 標準生成熱
Std Gibbs E of Formation KJ/mol 標準Gibbsエネルギー
Porlarzability 分極率
Ionization Potential イオン化エネルギー
LUMO 最低空軌道
RDkit-識別子の作成
RDKitは普通はPythonを使って利用するのが普通ですが、RDKitのJavaScript版を使いブラウザーから計算できるようにしてあります。
作成方法はYMBと同じで、Smilesをペーストして計算を繰り返し、最後に計算結果をテーブルに戻します。

とても簡単にテーブルを用意できるので、これについては自分でやってみましょう。
KDKitに関しても、リスト上のSmilesを一括で計算するProバージョンもあります。
ProバージョンはRDKitが生成した分子の3次元構造からCNDO/2分子軌道計算も行い、計算結果を一覧表にして返します。
CNDO/2, 分子軌道計算による識別子の作成
RDKitの機能を使うとSmilesの構造式から、3次元の分子座標を入手する事ができます。
そして以下のページでCNDO/2計算を行います。
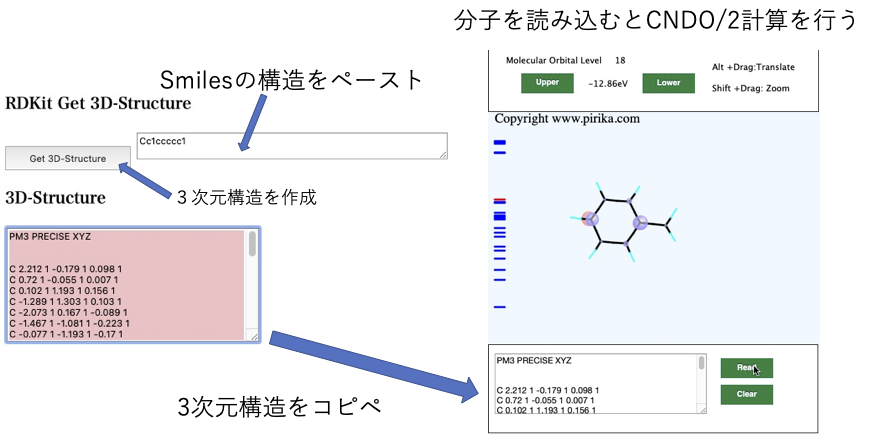
分子軌道法を使った識別子の作成は最もよく行われている方法です。
一応CNDO/2で計算した結果を添付しておきますが、良好な推算式の作成には至っていません。
ただ、今の時代ならCNDO/2で計算する必要はありません。RDKitで得た3次元構造をBabelソフトを使って、GAMESSなどのフォーマットに変換して、もっと高度な分子軌道計算にトライするのも良いと思います。
これができたら
その3、データ解析
へ進みます。
情報化学+教育 > MAGICIAN 養成講座 > マイクロ波と3つのMI > 有機物の識別子の作成法
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください) メールの件名は[pirika]で始めてください。
