MIRAI(Multiple Index Regression for AI)の新しいファミリーを使って、モデル式を作ってみた。
元データは国立医薬品食品衛生研究所の「人皮膚モデル(3次元皮膚モデルEPISKIN)を用いた皮膚刺激性試験代替法」という新規試験法提案書だ。
中身を理解しているわけでは無い。
今、pirika研究会で動物実験代替法をハンセンの溶解度パラメータなどから予測できないか、学生と始めようとしてしている。
そこで、ネットを調べていたら出てきたものだ。
ヨーロッパでは、動物実験禁止が進むので、こうした代替の試験方法や、機械学習を用いたQSARはとても重要になってくるだろう。
機械学習用の識別子としてハンセンの溶解度パラメータ(HSP)はとても重要だ。
所詮、皮膚を通じて体内へ侵入しなければ、毒も薬もない。
それ以外、logKow(オクタノール水分配比率), logS (水への溶解度), logBCF(生物濃縮性、HSPiPへはまだ未搭載)、RDKitのトポロジカルな情報を利用することができる。
混合物などは計算できないが、今回使った化合物のSmilesの構造式とDominant Medianの値を記載しておくので、我こそはと思う人はモデル式作成をチャレンジしてほしい。
この、Dominant Medianは自分は馴染みがないが、GHS分類でIrritantで2.3以上、Mild irritantで1.5以上2.3未満、non-Irritantで1.5未満となるらしい。
つまり、分子の構造のみから、Dominant Medianが予測できれば良い。
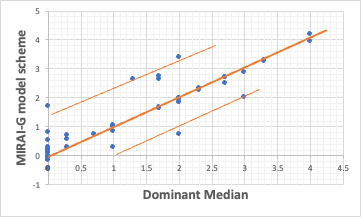
Pirikaで開発している解析ツールは、モデル式を構築する際に、誤差の取り方を4つの方法から選ぶことができる。
今回はMAEで解析を行うと上のような結果が得られた。49データのうち40データはほぼ綺麗にY=Xの直線に乗る。
そして上に偏位する化合物が5つ、下に偏位する化合物が3つ、あることがわかる。
つまり、MIRAI-GのMAEでモデル化すると、今回選んだ識別子で40化合物はよく記述できているが、もう2つ識別子が足りないことがわかる。
上に偏位する化合物、下に偏位する化合物がどんな特徴を持つ化合物なのか、考えることができる。
これが、中をブラックボックスにしてしまって、なぜだかわからないが全部平均的に線の上に乗ってしまうようなニューラルネットワーク法と大きく異なる点だ。
