2018. 12.21
情報化学+教育 > 情報化学 > フリー・ツールの利用 > SDF fileの取り扱い
Pythonとかプログラミングが使えないと困る事例の一つを、やはりブラウザーでやってみましょう。
それはSDFファイルの分割です。
pubchemなどから分子の構造と物性値などをダウンロードしてくると、sdfフォーマットでダウンロードされます。
(sdfファイルの詳細はこちらを参照してください。)
RDKitの中にも、化合物の水への溶解性の訓練セットと予測用セットが含まれています。
それを表計算用のソフトにペーストできるように、1行1化合物(物性値などはタブ区切り)になるように整形してみましょう。
RDKitのオリジナルをダウンロードして解凍すると、Docs/Book/dataの下にsolubility.test.sdfとSolubility.train.sdfの二つのファイルがあります。
もしかしたら、sdfフォーマットに関連づけられたアプリケーションがすでにあるかもしれませんが、textエディターで開いてみましょう。
最初から、M ENDの範囲は分子のmolフォーマットになっています。
3行目の6は原子の数、5は結合の数を示しています。
C言語の名残でしょうか、厳密に数字の位置が規定されているので、原子数が100を越えると原子の数と結合の数がくっついてしまい厄介な事が起きます。
また4行目からは原子の3次元座標と原子の種類が記載されます。
Z座標が全て0なのでこのmolファイルは2次元フォーマットである事がわかります。
そこでこのフォーマットでは分子軌道計算する用のデータには使えません。
また、水素は除外されています。その後からの
> <ID>
> <NAME>
> <SOL>
> <SMILES>
> <SOL_classification>が分子の情報になっていて、終わりの行に
$$$$があり次の分子に移ります。
こうした分子が1000分子以上まとまってTrain Data Setになっています。
これを手作業で表計算ソフトに落として行ってもいいのですが、自動的にやってしまいましょう。
次のページにアクセスすると下の画面が表示されます。

ここでは、まず区切りの文字列を入れます。
$$$$とM ENDは必須です。
それ以外の文字列は、分解したいSDFファイルをよく見て入力します。
できれば、SDFファイルからコピー/ペーストすると良いでしょう。
smilesと表記されたりSMILESと大文字になっていたり、ケースバイケースです。
取り出したくない文字列は入れなくても良いです。
> <などの制御文字や>の後ろに文字列があってもそれは無視してください。
準備ができたらファイルを選択ボタンを押します。
この機能はHTML5のFile APIを利用するので、この機能が搭載されていないブラウザーを利用した場合には動作しません。
読み込みに成功すれば1行1分子のテキストが出力されますので、全てを選択して表計算ソフトにペーストしてください。
ファイルの分割が失敗する大きな原因は、sdfファイルの改行コードです。web標準の”\n”でない、”\r\n”(Windows改行コード)などを使っていないかチェックしてください。
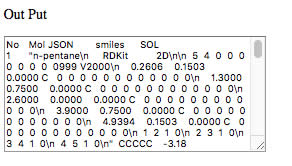
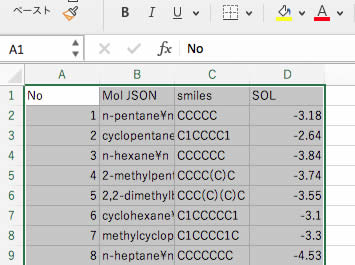
こうした物性値とSmilesのペアが簡単に手に入ります。
分子のMolファイルは本来は数行にわたるテキストデータです。
そのままでは表計算ソフトにはペーストできませんが、JSONという複数行を1行に変換する機能を使って貼り付けてあります。
ここでのmolファイルは2次元フォーマットで使い道は余りありませんので消去してしまっても良いです。
どのみち、SmilesをRDKitを使って処理する際には、3次元のMolファイルを発生させるので、そちらを使えば良いわけですから。
こうして、訓練用のデータ、テスト用のデータを準備して、訓練用のデータで機械学習を行い、テスト用のデータで精度を調べるのがケモ-インフォマティクス手法になります。
水への溶解度の推算式は、もっと少ないデータから構築して見ました。
(RDKitの分子への水素付加に間違いがありました。分子結合インデックスが間違っているので、結果も変わります。)
その時のScheme(2)は次のようになりました。
logS=-0.2183895636547375*Chi0n+1.1113943882657598*FractionCSP3+-0.6154433219798137*Kappa1+1.0678749094594537*LipinskiHBD+0.5755342165654573*NumHBA+-1.5559623499363546*Chi4n+-0.45241634258826235*LipinskiHBD*NumHBA+0.08721946861665164*Chi0n*Chi4n+1.9585849028724098 (2)この式を使って訓練用のデータを評価して見ましょう。
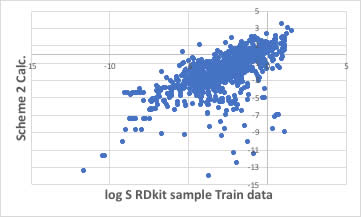
143個のデータから作った割には、そこそこの値になっています。
そこで機械学習やPLS回帰を使ってもっと良い式を組み立てて行くことになります。
PLS法で行なった例をこちらに示します。
もしくは、識別子の意味をもっと深く考えたいなら、値が外れる化合物を前回のテーブルに継ぎ足して変数選択重回帰を行えば良いのです。
SDFファイルは様々なデータセットのものがネットに転がっています。腕試しに色々遊べますので試して見てください。
EPAのHPからデータをダウンロードして解析する例題を付け加えました。
自分でそう言いながら、環境関連のデータセット(米国環境庁:EPAのT.E.S.T.データ)を落としてきて解析しようとして困りました。
このsdfデータには, Smilesの構造式が記載されていません。
CAS番号が記載されているので、
PubChem Identifier Exchange Service
などで一括変換するのが一つの手です。
このSDFファイル取り扱いソフトに、OpenBabelを組み込んで、MolファイルからSmilesを発生させるように改良してみました。
そこまでやるなら、実はRDKitも使って識別子も同時に発生させることも簡単にできます。
それはPro版に搭載しようと思っています。ました。
情報化学+教育 > 情報化学 > フリー・ツールの利用 > SDF fileの取り扱い
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください) メールの件名は[pirika]で始めてください。
