2024.7.28
pirika.comで化学 > 化学全般
> 次世代HSP2, Pirika Pro for MI > 複数の官能基を持つ場合の混合則
> ハンセン溶解度パラメータ (HSP) Doc
注意:HSPiPに搭載の機能ではありません
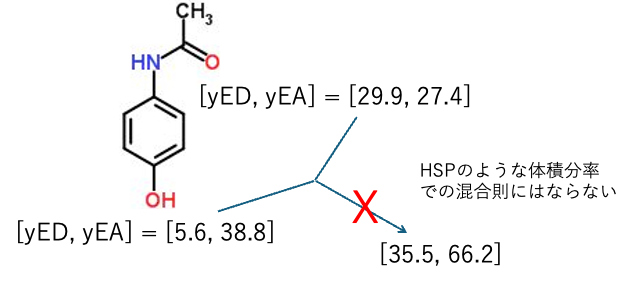
Pirika24Pro4MIにはYamamoto法のElectron Donor/Acceptorの推算式が搭載されている。詳しい推算法の構築に関しては「無機物の分散に酸性、塩基性は影響するだろうか?」を参照していただきたい。
原子団にはyED,yEAの値が割り振られている。そこでアミド基とフェノールのyED, yEAの値は抜き出すことはできる。しかし、HSPのようにその体積分率での和が分子としてのyED, yEAになるわけではない。
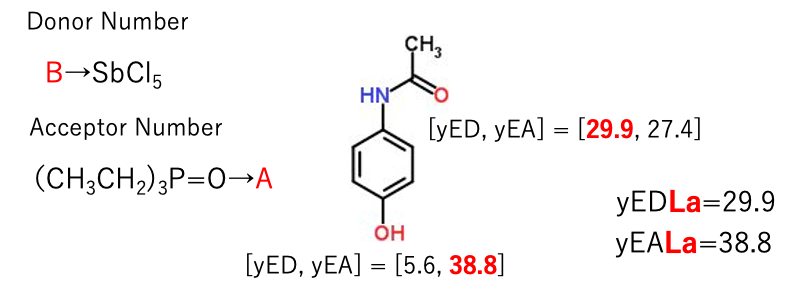
むしろ、分子中の一番大きなBがSbCl5へDonorする、一番大きなAがP=OからAcceptorすると考えたほうが合理的なような気がする。
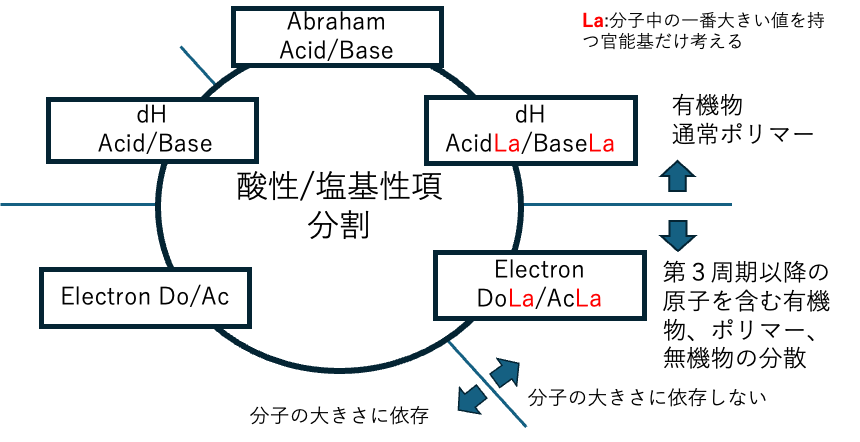
AbrahamのAcid/Baseは分子の大きさには依存していない。その値をハンセンの水素結合項に割り振ったdHAcid/dHbaseやそのLaを考えると5パターンある。(そもそも分ける必要がないdHのまま使う事もある)
分割された係数の取り扱いにも、主に2つのパターンがある。
Euclid Type式
(A1-A2)2+(B1-B2)2
Beerbower Type式
Coeff*(A1-A2)*(B1-B2)
Euclid Type式では交換作用は加味されない。一番高い相互作用を示すのは、A1=A2, B1=B2の時になる。
カルボン酸はA1, B1の値を持ち、分子内水素結合を作る。1種の塩になる。同じ分子同士なのでBeerbower Type式で考えても、係数はゼロになる。
しかし、弱酸の塩に強酸を加えれば交換する。つまりA2がA1より大きいか、B2がB1より大きければ式の値は負になる。そこで片っ端から距離の式を作り、溶解度との相関を検討してしまう。
Paracetamolの溶解度は「アセトアミノフェンの定量的溶解性」で提供しているので、MIツールを持っているなら自分で計算してみよう。
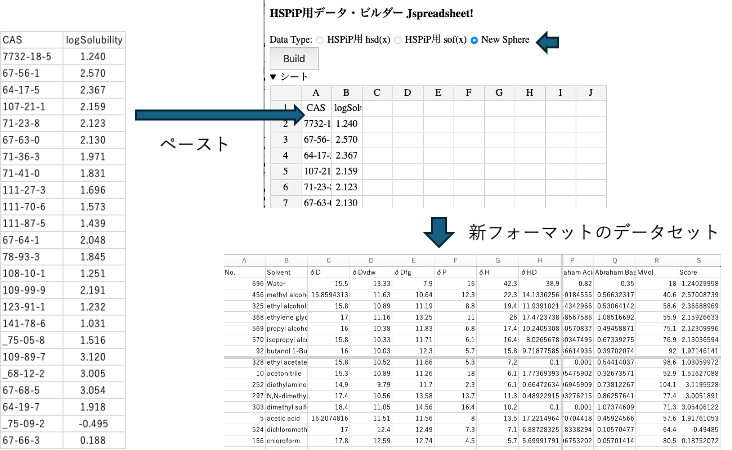
CAS番号と溶解度のデータをデータ・ビルダーWebアプリにペーストする。
New Sphereを選択してBuildボタンを押すと、新しいフォーマットのデータセットが作成される。(ここでHSPiP用のhsdxを指定すればHSPiPですぐに読み込める)
WebアプリはCAS番号を調べ、オフィシャル値があればそれを用い、なければYMBで推算した値を集めテーブルを作る。CAS番号が登録されていない場合には、SMILESの構造式を用意し、YMBのWebアプリを使ってデータセットを作る。
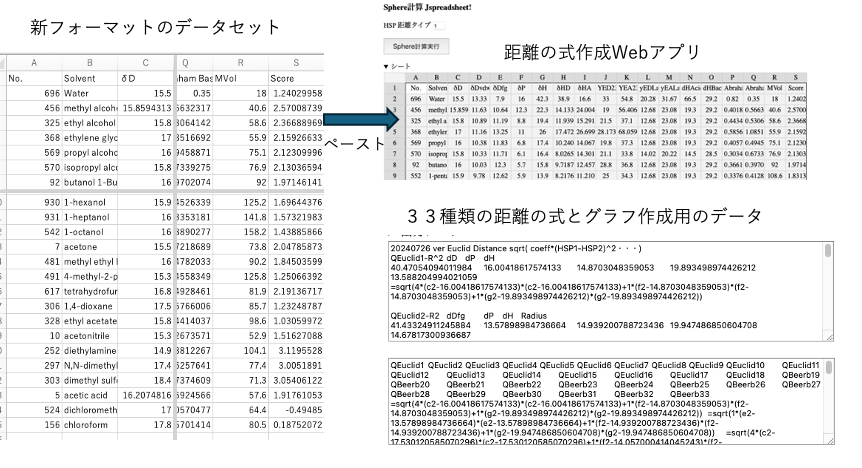
計算には少々時間がかかる。
計算が終わるとBeep音が鳴るようにしてあるのでゆっくり待とう。
距離の式とグラフ作成用のデータをExcelに戻しておく。距離の式の意味は
「新しいHSP距離の考え方 量子ドットを例に」で詳細に解説している。
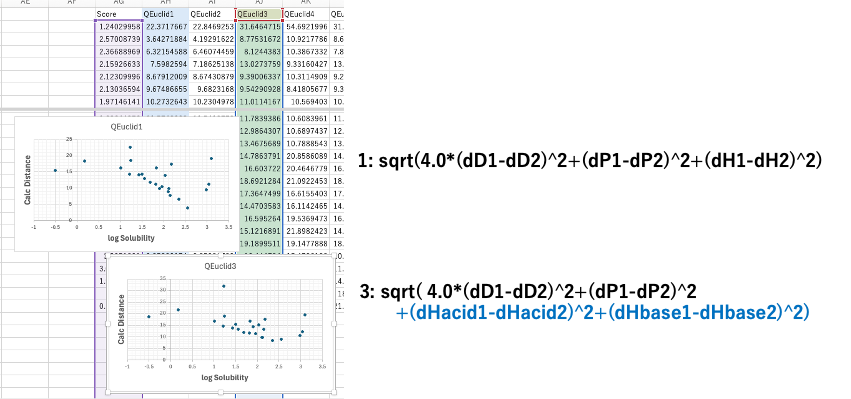
結果は簡単に図示することができる。各距離の式と溶解度の相関係数を算出してある。どの式が最も適しているかは相関係数から判断するのも良いが、相関係数は一つ大きく外れるものがあると極端に悪くなる。丁寧にグラを見ながら判断するのが良いように思える。
上図のEuclid Type 1,3はHSPiPに搭載されている。Data pointsを使えば似たようなグラフが得られるだろう。ほとんどは線の上に乗っているのだが、特に溶解度が大きなところで大きくばらつく。そこで、溶解度の高いところで安定な値を与える距離の式を探す。
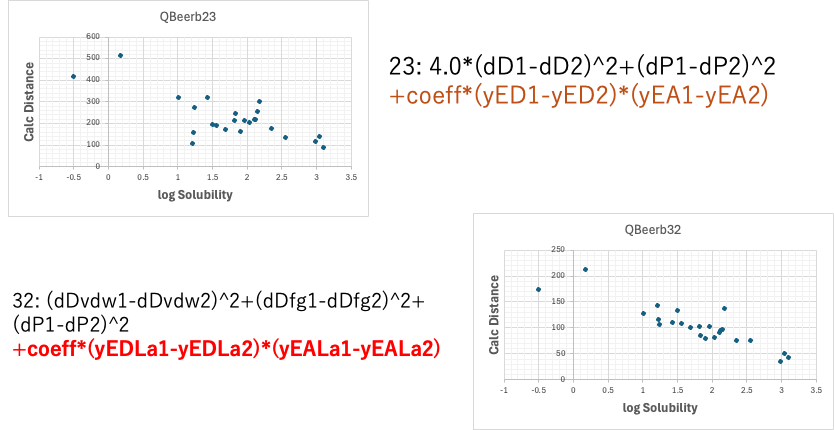
Euclid Typeの式では皆ばらつくが、Beerbower Typeの式では式番号32がとても良い結果となった。
これを勝手にデータ駆動型研究と偉そうに呼んでしまう。
このParacetamolの溶解には、分子中の最も大きい、yEDLaとyEALaがBeerbower Typeの交換安定型の相互作用が働いていると解釈すると、理解しやすい。
ただし、さまざまな系で計算してみると、何時もそうなるとは限らないようだ。
この場合はParcetamolが複数の官能基を持っていたことが大きい。
そこで、やはり、混合則と呼べるほどの規則は、やはり、まだ見つからない。
当面は、片っ端から計算して、データを集めるしかなさそうだ。
しかし、MI的に有用な式を作成し、とりあえず良いものを特許取るという方針であるなら、使わない手はないだろう。
ただし、Beerbower型の距離の式は、ハンセン球の中心といった明確な距離を最小にする点は無い。
使える(使いたい)溶媒に対して片っ端から得られた式で距離を計算して一番短くなるものをスクリーニングする必要がある。その辺が少し面倒だ。
データの作成法、距離の式の求め方を説明
書いて説明するのは難しいのでV-tubeを作った。
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください
