HSPiPの概要 | GUIを用いた物性推算ソフト、Y-Predict
2012.Dec.19
Y-Predictは熱力学的物性値を推算するプログラムです。物性推算の基礎式はwww.pirika.comで開発されたものですので、詳しい説明、予測精度に関してはHPを参照してください。
HSPiPに搭載されているY-MBは、分子構造の入力はSmilesの構造式、InCheの構造式、MOLファイルからの入力のみで、GUIでの入力には対応していません。そこで、HTML5ベースのGUIソフトウエアー、Y-PredictをHSPiPのパワーツールとして提供いたします。
Y-PredictはHSPiP を購入されてライセンスファイルをお持ちの方だけがお使いいただけます。
ブラウザーの対応状況:
| Mac | Windows | Linux | |
| Chrome (ver. 23) | ◯ | ◯ | |
| FireFox (ver. 17) | 一部制限(Web Storge X) | ◯ | |
| Safari(ver. 6以上) | ◯(OSX, Lion+) | ? |
|
| Opera (ver. 12) | X (File API使えない) | ◯ | |
| IE (ver. 10) | X (Mac版なし) | 一部制限(Web Storge X) |
インターネット・エクスプローラー(IE)のver.6から8はHTML5に対応していない為、動作しません。IE-9はFile APIに対応していないため利用できません。IE-10はLocal Storgeに不具合があるため、毎回認証を行わなければなりません。マック版のOperaはFile APIが使えないため認証が行えません。Mac版のFireFoxもLocal Storgeが消えてしまうことがあり、その場合は認証をやり直さなければなりません。
一番安定して使えるのはChromeブラウザーです。Linuxn用のブラウザーは確認がとれ次第情報をアップデートしていきます(情報をお持ちの方、フィードバックを頂けるとありがたいです)。
分子の描画についてはDraw2Smilesと同じなので、画面はそちらを流用しています。
スタートアップ画面
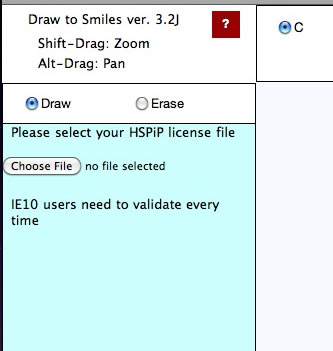
初めて使う場合には、上記の画面が現れます。(画面はブラウザーによって、ボタンの名前、見かけが異なります。)パワーツールを使い始める前に、HSPiPのライセンスを持っていることをブラウザーに教える必要があります。”ファイルを選択ボタン”を押して、HSPiPのライセンスファイルを指定します。(Windowsで利用の場合は、Program Files¥Hansen-Solubility¥HSPiP¥XXXXXX.licenseにあるはずです。)
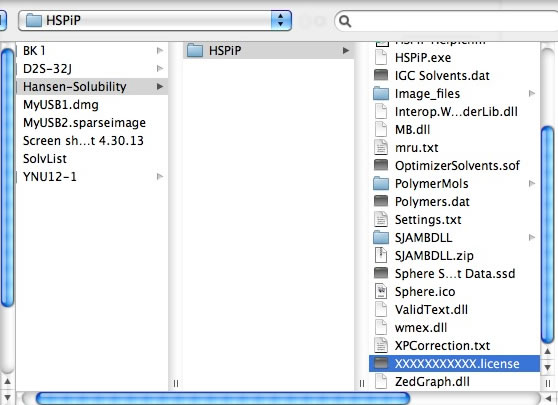
一度、ライセンスファイルを開き、それが正しいライセンスと認識されると、ブラウザーのLocal Storge(クッキーのようなもの、HTML5から導入されています)に登録され、以後はどのパワーツールを使う場合でも認証なしに利用できるようになります。
Mac版のSafari (ver. 6未満), OperaとWindow版のIE-9はライセンスファイルを選択しても、ファイルの中身を読み込む機能が無い(File APIの実装が不十分)ため、認証できません。またIE-10では読み込み、認証はできるのですが、Local Storgeで読み込んだ結果を保持することができないので、パワーツールを走らせるたびに認証を行う必要があります。Mac版のFireFoxもLocal Storgeが消えてしまうことがあり、その場合は認証をやり直さなければなりません。
認証が正しく行われると、以下の画面になります。
薄い水色の部分はキャンバスと呼ばれる領域で、描画はこの領域に対して行われます。ボタン、ラジオボタン、テキストなどはHTML5+CSSで整形されるため、HTML5に未対応のブラウザーでアクセスした場合には表示はめちゃくちゃに崩れます。ブラウザーのサイズを変えると自動的にキャンバスのサイズは調整されます。
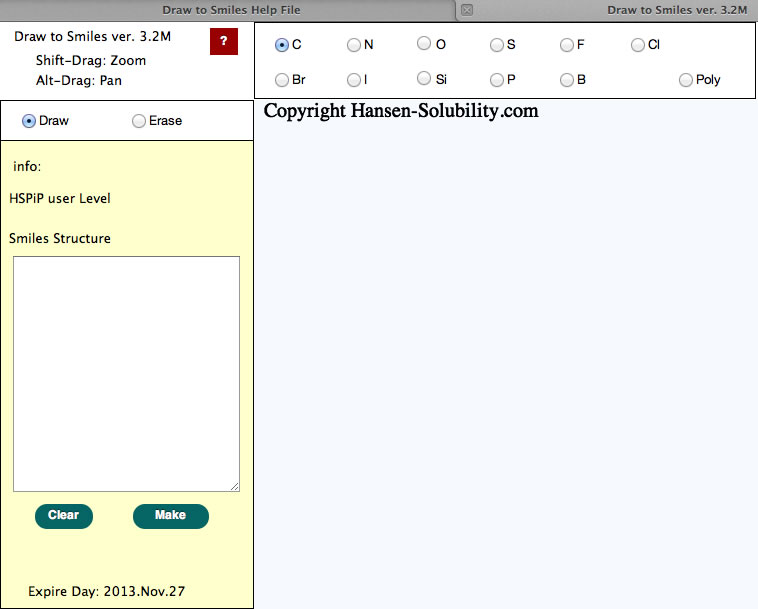
ラジオボタンでは、描画する原子の種類、描画モード、消去モードを選択することができます。
キャンバスの中でマウスボタンを押すと水紋のような模様が現れます。マウスボタンを押したままマウスを動かすと結合が現れます。結合は15度ごとの飛び飛びの値になります。
 |
 |
マウスボタンを離すとラジオボタンで選択した原子が結合の両端にプロットされます。(マウスを動かさないで、押して離すと1つだけプロットされます。)(タッチパネル・デバイスでは動きませんが)マウスのボタンは押さないで片方の原子の上に合わせると、下の絵のように原子の周りに水紋が現れます。これはプログラムが、そこに原子があると認識した状態です。水紋が現れた状態でマウスボタンを押して、押し続けたままマウスを動かす(ドラッグする)と新しい結合が作られます。分子が大きくなると原子の認識に時間がかかります。あまり早く操作すると新しい原子をその場所にプロットしてしまうので注意してください。
ラジオボタンで他の原子を選択した場合、以後、マウスボタンを離した所には新しい原子がプロットされます。
既に描画済みの原子の上で(水紋が出てから)マウスボタンを押して、すぐ離すとその原子を置き換えることができます。
2重結合を描画したい場合には、マウスを最初の原子の上に合わせて(水紋が現れたら)、マウスボタンを押し、2重結合を作りたい原子の上まで持って行き(水紋が現れたら)ボタンを離します。
 →
→
消去モードを選択すると、キャンバスの色が変わります。(これはモードが変わっていることを思い出すため)
消去モードでマウスを原子の上に合わせ、水紋が現れた後、マウスボタンを押して離すと原子が削除されます。原子と原子の中間にマウスを合わせると、両方の原子に水紋が現れます。そこでボタンを押すと結合が消去されます。
 →
→
Altキー(MacではOptionキー)を押しながらドラッグすると分子を移動することができます。一つの原子の上で(水紋が出てから)Altドラッグするとその原子だけを移動することができます。 混みあった分子を描く時に重宝します。
 →
→
Shiftキーを押しながら左右にドラfッグすると拡大、縮小することができます。
 →
→
(注意:キー操作を併用するする場合、マウスボタンを離してから、キーを離してください)
分子が出来上がったら、推算(Predict)ボタンを押します。テキストエリアに推算結果が表示されます。また2次元QEQ(説明はPirika.comのHPを参照)を使った原子電荷が表示されます。必要がなければチェックボックスのチェックを外せば表示はしません。(その際に自動的に水素を付加して計算しますが、水素は表示されません。表示して欲しいというフィードバックが多ければ、優先順位を上げて作業します。)
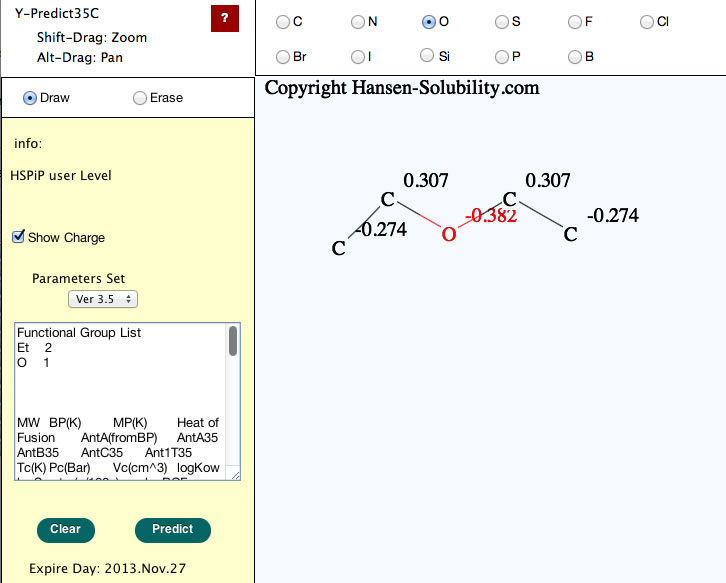
官能基のリストを良く見て、間違った分割をしていないかチェックしてください。もし間違った認識をしていたら是非ともフィードバックをしてください。
テキストエリアの計算結果(必要に応じてタイトルも)を選択してコピー(Ctr-C)してください。

結果をスプレッドシートにペーストすれば、後は自由にアレンジすることができます。
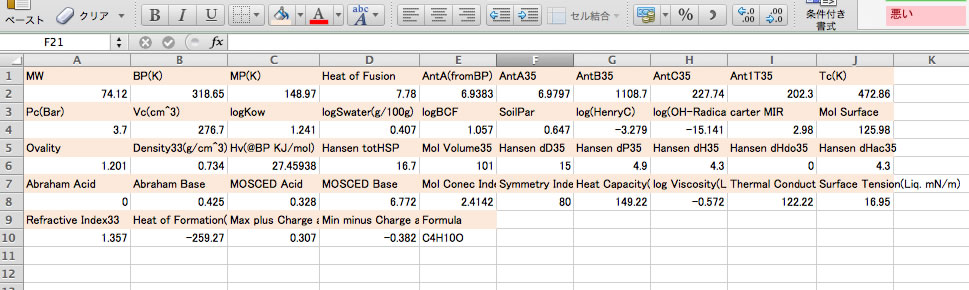
Ant1T35:蒸気圧が1mmHgになる温度(K単位)
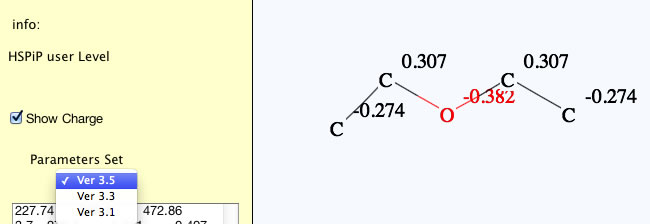
パラメータセットは、3.5, 3.3, 3.1がプルダウンメニューから選択できます。以前の計算結果のほうが自分にとってよく合っていたなどの場合にはそちらをお使いください。(HSPiPのセットは ver. 3.1です)
ニトロ化合物は便宜的に以下のように表記してください。
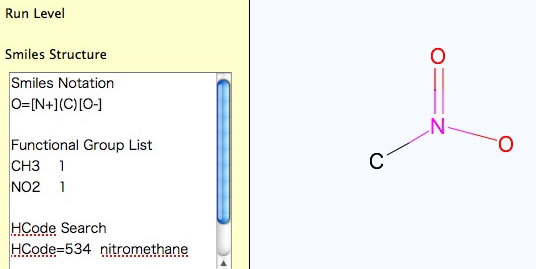
なれるまでは、ゆっくり操作してください。特に大きな分子になってくると、マウスの位置に原子があるかどうか判定に時間がかかります。認識する前に手を離すと思わぬ所に原子を付け加えてしまいます。
使える最大原子数は重原子120(HSPiPと同じ)ですが、推算の精度が意味を持つのは、沸点にして700Kぐらいの化合物です。それより大きな分子は実験値が無いかあやふやになるので信頼できません。
大きな分子の物性で意味のあるデータは、HSP, 融点とlogP、水への溶解度ぐらいなので、次期バージョンアップでは分子のタイプごとに推算式を切り替えることが検討されています。
新バージョン(2013.8.15)では物性値の温度依存性を計算するようになりました。
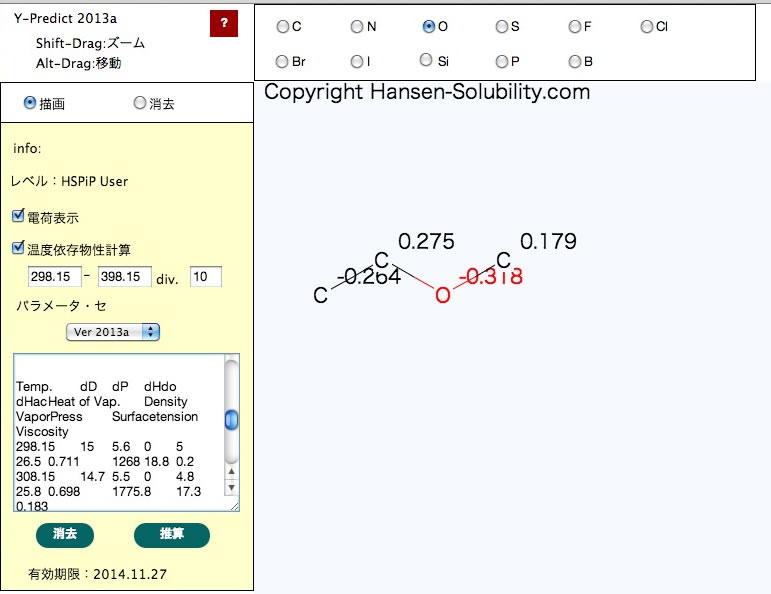
開始温度と終了温度、そして温度の刻み幅を入れて、チェックを入れると推算ボタンをクリックした時に、HSP, Heat of vaporization, density, Vapor pressure, Surface tension そして viscosityを計算します。
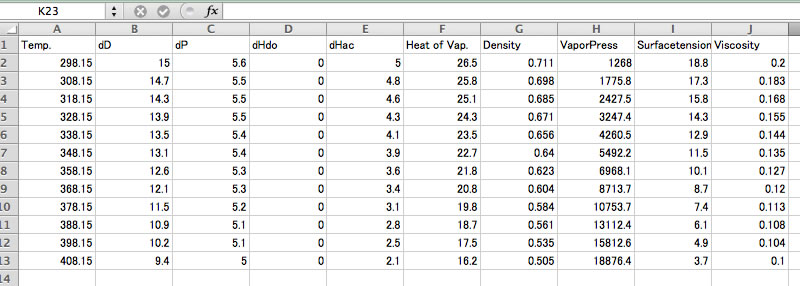
計算結果はコピーして表計算ソフトなどにペーストして利用することができます。
注意:HSPの温度依存性はHSPiPに搭載されている、熱膨張係数から求めるやり方とは全く異なっています。各温度での蒸発潜熱と分子体積を算出して、tot HSP(T)=sqrt((Hv(T)-RT)/V(T))からトータルHSPを求め、25℃での標準のHSPが正しい値になるように、dD, dP, dHdo, dHacの温度依存性を求め決定しています。