2005.7.20(2024.9.9改訂)
pirika.comで化学 > 化学全般 > 高分子化学 >
CNDO/2でモノマーの計算
2024年現在、CNDO/2の計算はSMILESの構造式から行うようにしている。
そこでモノマーのSmilesの構造式は集めておく必要がある。
それさえ準備できていれば、つぎのWebアプリにSMILESの構造式を貼り付けてReadするだけで計算できる。
例えば自分が使いそうなモノマーを次のようにテーブルを作っておけば良いだろう。SmilesをコピペすればCNDO/2を計算することができる。
| CAS# | Name | Smiles |
|---|---|---|
| 79-06-1 | Acrylamide (Ethylenecarboxamide) | C=CC(N)=O |
| 79-10-7 | Acrylic acid | C=CC(O)=O |
| 107-13-1 | Acrylonotrile | C=CC#N |
| 591-87-7 | Allylacetate | CC(OCC=C)=O |
| 107-18-6 | Allyl alcohol | C=CCO |
| 107-05-1 | Allyl chloride | C=CCCl |
| 106-98-9 | 1-Butene | C=CCC |
| 141-32-2 | Butyl Acrylate | O=C(C=C)OCCCC |
| 97-88-1 | Butyl Methacrylate | C=C(C)C(OCCCC)=O |
| 5976-47-6 | 2-Chloroallyl alcohol | C=C(Cl)CO |
| 557-98-2 | 2-Chloropropene (2-Chloro-1-Propene) | CC(=C)Cl |
| 563-47-3 | 3-Chloro-2-methylpropene | CC(=C)CCl |
| 513-37-1 | Methallyl chloride | Cl\C=C(/C)C |
| 683-51-2 | alpha-Chloro acrolein | Cl/C(=C)C=O |
以降、2005.7.20の古い記述。
CNDO/2でモノマーを計算する場合は[こちらのソフトをご利用]()ください。モノマーの構造はJSMEを使って描画します。
運良く古いJAVAのアプレットが動けば、次の画面が現れる。(現れなかったら諦めてください。)

画面はMacのSafariブラウザーで動かしたもの。(2011.11.22)
骨格となるモノマーをプルダウンメニューから選び、Searchボタンを押す。この部分はJavaScriptを使っているので、JavaScriptの機能がOffになっていると動作しない。
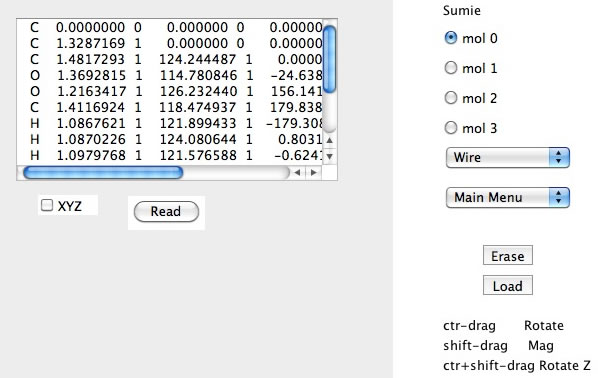
するとJAVAScriptの見つけた構造がJAVAのプログラムに引き渡される。この画面から先はJAVAのプログラムになる。分子構造を見て、このデータは内部座標系なのでXYZのチェックが外れていることを確認してReadボタンを押す。
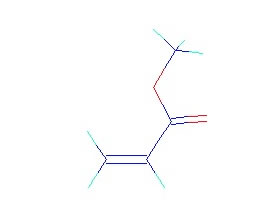
すると分子を描画する。
メニューからFast Buildを選ぶ。
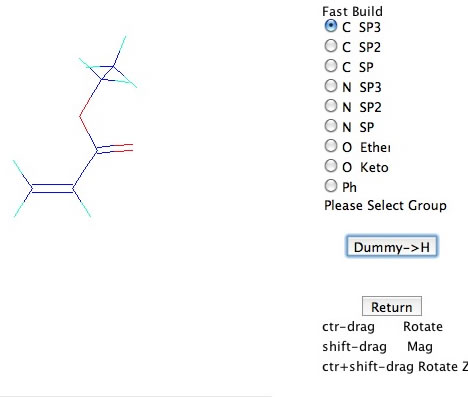
ここで、原子団のタイプをラジオボタンから選ぶ。そして先に元になる原子を選び(赤丸が表示される)次に切り離す原子を選ぶ。するとそこに選んだ原子団が付加される。そしてダミーアトムを水素に変えると上のような分子構造が得られる。
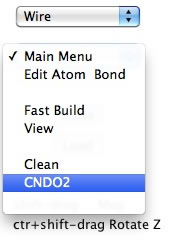
そしてメニューからCNDO/2を選択する。
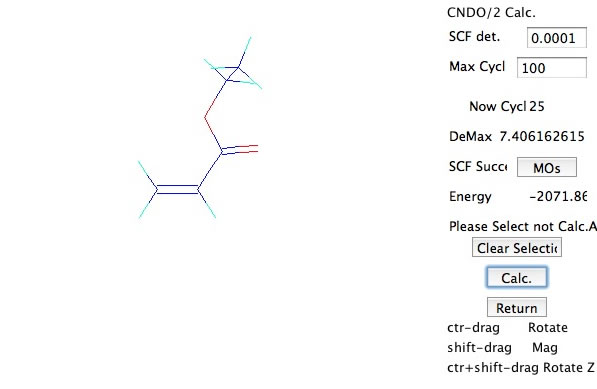
そしてCalc.ボタンを押すとCNDO/2を計算する。成功したらMOsのボタンを押す。
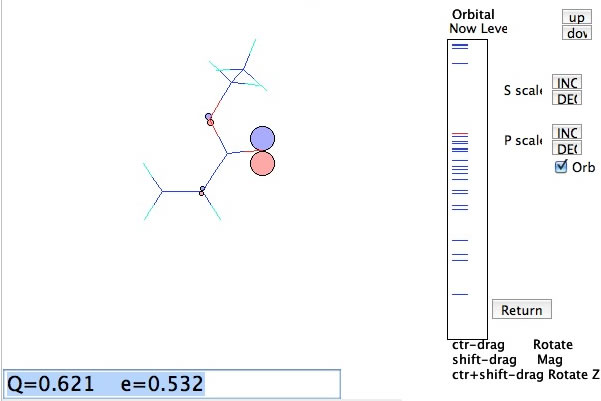
自動的にHOMOの分子軌道図を描く。levelのup downで他の起動を見ることができる。
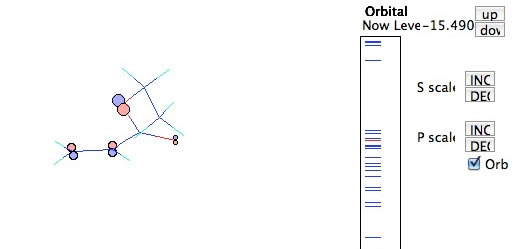
2重結合の結合性のπ軌道はHOMOより低いところに来ている。必要があればP軌道のスケールを上げていけば見やすくなる。ラジカルは2重結合のπ軌道の大きい方にアタックする。大抵の場合、Tail(尾)-すいている方の炭素に反応し、Head(頭)-原子団が付いている方の炭素が次のラジカルになる。
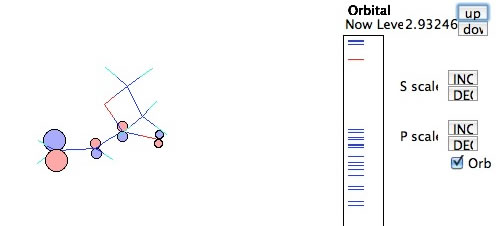
反結合性のπ軌道はLUMOで有ることが判る。
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください。
