

| Home | 化学 | HSP | 情報化学+教育 | PirikaClub | Misc. |
| 化学トップ | 物性化学 | 高分子 | 化学工学 | その他 |
2005.1.14
この事は教科書的にはラジカルのSOMOとモノマーのHOMO,LUMOで決まると言われているこの反応には何か他の因子が絡んでいることを示唆しています。
たとえばラジカルのheadの炭素の電荷とかLUMOのエネルギーレベル、モノマーのTailの炭素の電荷とかでしょう。考えつくすべての因子を入れて主成分分析によって絞り込んで行くのも一つの手です。自分はラジカルのLUMOのエネルギーレベルがこの反応に関与していると考えているのでラジカルのSOMO,LUMOとモノマーのHOMO,LUMOでもう一度ニューラルネットワークを組み直してみます。
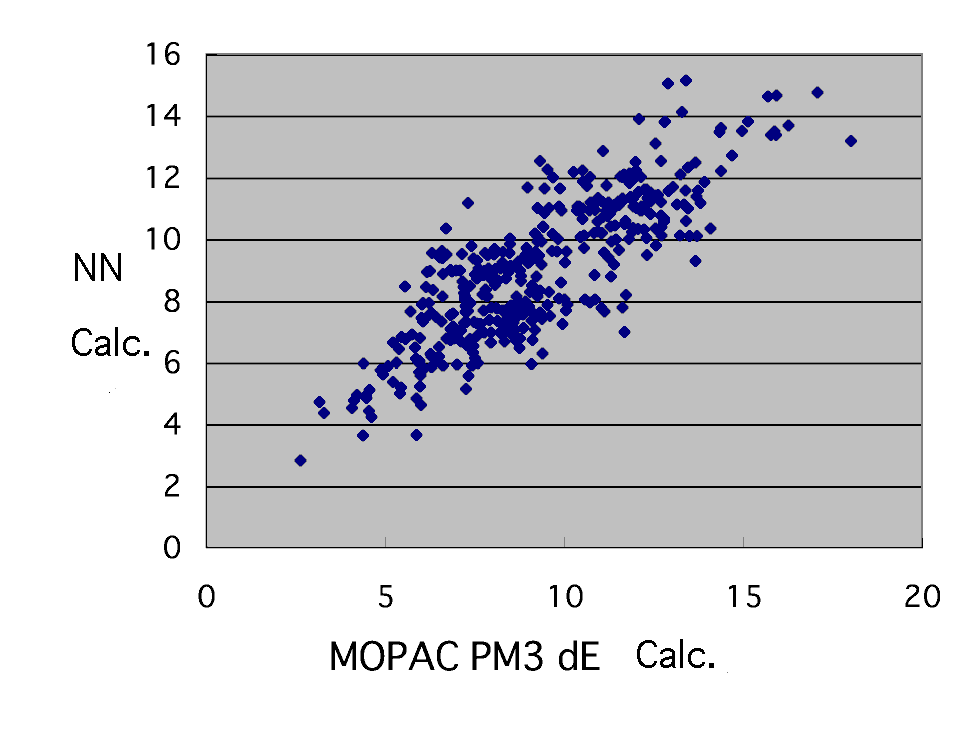
中間層の数を5としてバイアスニューロンありで490000回学習(再構築学習法)したところ次のような結果になりました。

重回帰と比べれば随分改良されたのが判るとおもいます。次に何をしたらいいでしょうか? 中間層を変えてみる、電荷を入れてみるなど色々やれる事は一杯あります。ついついもっと精度の良い直線にびたーっと乗ったグラフを書きたくって先を急ぐのもよく判ります。しかし、もしニューラルネットワーク法を身につけたいと思われるのでしたらちょっと踏みとどまってください。
ここではまずどんな系が直線からずれているかチェックしてみます。
| name | dE | NNの 答 え | 誤差 の 絶対値 | RaSOMO | RaLUMO | MonHOMO | MonLUMO |
| cHn-AMD | 11.68 | 7.69 | 3.99 | -8.895 | 1.047 | -9.794 | 0.143 |
| AA-MeO | 10.48 | 14.33 | 3.85 | -8.803 | 0.160 | -9.515 | 1.302 |
| AMD-AA | 6.70 | 10.53 | 3.83 | -9.647 | -0.480 | -11.147 | -0.170 |
| AMD-MeO | 7.91 | 11.67 | 3.76 | -9.647 | -0.480 | -9.515 | 1.302 |
| BD-cHn | 18.01 | 14.46 | 3.55 | -8.848 | 0.211 | -9.593 | 1.168 |
| Mal-MeO | 5.27 | 8.68 | 3.41 | -10.963 | -1.509 | -9.515 | 1.302 |
| MAN-MeO | 11.29 | 14.69 | 3.40 | -9.486 | -0.617 | -9.515 | 1.302 |
| VFR-AMD | 7.26 | 3.96 | 3.30 | -9.343 | 0.704 | -9.794 | 0.143 |
| Vac-Mal | 7.31 | 10.61 | 3.30 | -8.785 | 0.553 | -11.708 | -1.548 |
| VDF-MeO | 4.15 | 7.24 | 3.09 | -9.874 | 0.113 | -9.515 | 1.302 |
| MeO-MeO | 7.35 | 10.41 | 3.06 | -8.352 | 1.433 | -9.515 | 1.302 |
| MMA-MeO | 11.68 | 14.74 | 3.06 | -9.566 | -0.598 | -9.515 | 1.302 |
| AN-MeO | 7.84 | 10.89 | 3.05 | -9.855 | -0.719 | -9.515 | 1.302 |
| AMD-GMA | 6.72 | 9.71 | 2.99 | -9.647 | -0.480 | -10.587 | 0.143 |
| VFR-cHn | 10.00 | 7.06 | 2.94 | -9.343 | 0.704 | -9.593 | 1.168 |
| Vac-MeO | 7.71 | 10.63 | 2.92 | -8.785 | 0.553 | -9.515 | 1.302 |
| AMD-Vac | 6.31 | 9.21 | 2.90 | -9.647 | -0.480 | -10.083 | 0.687 |
| cHn-AllC | 11.03 | 8.14 | 2.89 | -8.895 | 1.047 | -10.269 | 0.621 |
| cHn-CHO | 11.62 | 8.73 | 2.89 | -8.895 | 1.047 | -10.681 | -0.098 |
| MAA-MeO | 11.20 | 14.07 | 2.87 | -9.682 | -0.717 | -9.515 | 1.302 |
MOPAC PM3の計算によるdEに対してニューラルネットが返す答えが大きくずれるものをエクセルで並び替え機能を使ってみてみます。そうするとワースト20のうち10個がメチルビニルエーテル(MeO)モノマーの系である事がわかります。しかも常にニューラルネットワークが3kcal/mol程度高い値を予測しています。このような事はどのようなときに起きるかと言えばMeOモノマーの生成熱がさらに3kcal/mol程度低い構造が存在することを示唆しています。(各遷移状態が3kcal/mol高い構造が存在していても同じですが、遷移状態は一番低いものだけが意味があって高いところにある遷移状態は通らないので関係ない。)
そこでメチルビニルエーテル(MeO)の構造を見てみると、
この図の上のような構造をとっています。エーテル酸素の周りは自由回転なのでぐるりと回して下のような構造を取ってやると1.607kcal/mol安定でした。そこでdEのテーブルをMeOの行だけ全部修正します。HOMO,LUMOの値も微妙に変わりますのでこれも全部修正します。そして新たに作り直したデータでもう一度ニューラルネットワークを計算します。このように修正しては計算、修正しては計算を延々繰り返します。
ここでMeOを修正すると次はこのようになります。
| name | dE | NN 推算値 | 絶対 誤差 | somo | lumo | MonHOMO |
| BD-cHn | 18.01 | 13.17649712 | 4.833502884 | -8.848 | 0.211 | -9.593 |
| cHn-AMD | 11.68 | 6.985349722 | 4.694650278 | -8.895 | 1.047 | -9.794 |
| BD-AMD | 13.66 | 9.284698015 | 4.375301985 | -8.848 | 0.211 | -9.794 |
| Vac-Mal | 7.31 | 11.17093357 | 3.860933572 | -8.785 | 0.553 | -11.708 |
| cHn-CHO | 11.62 | 7.787294954 | 3.832705046 | -8.895 | 1.047 | -10.681 |
| MMA-AMD | 14.09 | 10.33804494 | 3.751955055 | -9.566 | -0.598 | -9.794 |
| AMD-AA | 6.7 | 10.33514547 | 3.635145471 | -9.647 | -0.48 | -11.147 |
| MAA-AMD | 13.71 | 10.1018309 | 3.6081691 | -9.682 | -0.717 | -9.794 |
| cHn-AM | 11.72 | 8.193541698 | 3.526458302 | -8.895 | 1.047 | -11.067 |
| cHn-MMA | 11.12 | 7.658936543 | 3.461063457 | -8.895 | 1.047 | -10.553 |
| GMA-AMD | 13.5 | 10.1030179 | 3.396982101 | -9.699 | -0.74 | -9.794 |
| cHn-AllC | 11.03 | 7.760870639 | 3.269129361 | -8.895 | 1.047 | -10.269 |
| AMD-Vac | 6.31 | 9.552774897 | 3.242774897 | -9.647 | -0.48 | -10.083 |
| Vac-MeO | 9.311734 | 12.52751168 | 3.215777684 | -8.785 | 0.553 | -9.619 |
| AA-AMD | 9.07 | 5.94931352 | 3.12068648 | -10.118 | -0.814 | -9.794 |
| BD-CHO | 13.23 | 10.1212422 | 3.108757801 | -8.848 | 0.211 | -10.681 |
| AM-AMD | 9.38 | 6.291948181 | 3.088051819 | -10.017 | -0.711 | -9.794 |
| Vac-AA | 6.59 | 9.610234461 | 3.020234461 | -8.785 | 0.553 | -11.147 |
| Vac-VDC | 5.55 | 8.46468061 | 2.91468061 | -8.785 | 0.553 | -9.744 |
| Vac-Vac | 6.53 | 9.404180911 | 2.874180911 | -8.785 | 0.553 | -10.083 |
| VDC-AMD | 12.31 | 9.490990594 | 2.819009406 | -8.803 | 0.16 | -9.794 |
どれが悪かったのかを見るとアクリルアミドのモノマーを使ったものが軒並み悪いという結果になりました。しかも今度はニューラルの結果が3kcal/mol低くなるという結果です。このニューラルの結果が正しいのだとすると遷移状態がもっと低いところにある。か、AMDモノマーの生成熱がもっと高いところにあるという事を示しています。しかしAMDモノマーの生成熱がもっと高いところにあるものは安定構造ではないので本来はそちらは考えません。しかし今回はMOPACのバージョンが7と97が混在するためその可能性も含めて検討してみましたが3kcal/mol低くなることはありませんでした。結論から言うとこの場合はアミドモノマーのHOMOの位置の取り違えがこの計算誤差につながった事がわかりました。
| Root No. | 12 | 13 | 14 | 15 | 16 | ||
| 12A | 13A | 14A | 15A | 16A | |||
| -10.925 | -10.786 | -9.794 | 0.143 | 1.62 | |||
| S | C | 1 | -0.0217 | -0.0922 | 0.0167 | -0.0071 | 0.2072 |
| Px | C | 1 | 0.0252 | 0.1019 | -0.0167 | -0.008 | -0.0712 |
| Py | C | 1 | -0.0574 | -0.2712 | 0.0426 | 0.0031 | 0.186 |
| Pz | C | 1 | 0.657 | -0.1608 | -0.0569 | 0.5128 | 0.1986 |
| S | C | 2 | -0.0022 | -0.0013 | -0.003 | 0.019 | -0.0901 |
| Px | C | 2 | -0.0144 | -0.0457 | 0.006 | -0.0186 | 0.0878 |
| Py | C | 2 | 0.0132 | 0.0701 | -0.0134 | -0.0036 | 0.0475 |
| Pz | C | 2 | 0.6367 | -0.1571 | -0.0652 | -0.6589 | -0.1594 |
| S | H | 3 | 0.017 | 0.0793 | -0.0135 | -0.0067 | -0.0308 |
| S | H | 4 | -0.02 | -0.1008 | 0.0161 | -0.0097 | 0.097 |
| S | C | 5 | -0.0038 | -0.0389 | -0.0204 | -0.0005 | -0.3267 |
| Px | C | 5 | -0.0086 | -0.0792 | 0.0425 | -0.1061 | 0.2679 |
| Py | C | 5 | 0.0225 | 0.089 | -0.0139 | -0.0753 | 0.1662 |
| Pz | C | 5 | -0.0868 | -0.0111 | 0.052 | 0.3687 | -0.2106 |
| S | O | 6 | 0.0041 | -0.0018 | -0.0001 | -0.0037 | 0.0315 |
| Px | O | 6 | 0.259 | 0.7406 | 0.015 | 0.1013 | -0.1683 |
| Py | O | 6 | 0.0315 | -0.3488 | 0.1241 | 0.0823 | -0.0908 |
| Pz | O | 6 | -0.2592 | 0.1421 | -0.4444 | -0.3015 | 0.138 |
| S | H | 7 | 0.0301 | 0.1215 | -0.0138 | -0.0194 | -0.0008 |
| S | N | 8 | 0.0484 | 0.1695 | 0.1169 | -0.0214 | 0.4984 |
| Px | N | 8 | 0.0941 | 0.1827 | -0.3405 | 0.0955 | 0.0597 |
| Py | N | 8 | 0.0089 | 0.0164 | -0.2064 | 0.0427 | -0.0007 |
| Pz | N | 8 | -0.0116 | 0.1789 | 0.7713 | -0.1682 | -0.0181 |
| S | H | 9 | -0.0273 | -0.1036 | -0.0452 | 0.0268 | -0.358 |
| S | H | 10 | -0.0331 | -0.0871 | -0.0438 | 0.0165 | -0.3695 |
電子が詰まるのはRootNoで14、ですからLUMOのエネルギーレベルは0.143はOKだと思います。ここでHOMOをRootNo.14の-9.794を採用していたのですがよく見るとこれはアミドの窒素のPz軌道のものでした。C=Cの2重結合の軌道はRootNo.12の-10.925の所にありました。これを修正する事に依ってAMDモノマーがあわないのが修正できました。
また、アクリル酸でもMeO同様にもっと低いエネルギーのモノマーが見つかってこれも修正しました。
これで系統的にずれるものはなくなったようです。この後は個別に遷移状態が変なものを探して行きます。
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください) メールの件名は[pirika]で始めてください。